Es empfiehlt sich, zunächst einen Lauf mit 0 Iterationen durchzuführen. So kann ohne großen Aufwand die Zielfunktion kontrolliert werden. Die Ergebnisse werden in die Ausgabedatei (hier: out.inv) geschrieben, dabei werden die Anteile pro Parameter/Messwertgruppe einzeln aufgeführt.
Wichtig ist, die Anteile der Zielfunktion ausgeglichen zu gestalten. Nach dem ersten inversen Berechnungslauf (0 Iterationen) können die Anteile der Zielfunktion miteinander verglichen werden. Die am besten bekannten Größen (meist Potentiale) sollten dabei den größten Anteil an der Zielfunktion (ZF) haben.
Mögliche Fehlerquellen:
K-Werte sind zu stark gewichtet. Damit ist es dem Algorithmus nicht erlaubt, den K-Wert weit vom Schätzwert zu berechnen, und es ergeben sich fast keine Änderungen in der Verteilung der Durchlässigkeiten.
Abhilfe: Standardabweichungen vergrößern
Mengen sind zu stark gewichtet. Somit verlieren die Messungen der Potentiale an Gewicht und der Algorithmus versucht (vergeblich!), die Mengen zur Übereinstimmung zu bringen. Meistens gilt die Regel: Stimmen die Potentiale und die K-Werte, sollten die Mengen sich auch einstellen.
Abhilfe: Anteile der Zielfunktion ausgeglichen gewichten (mittels gut gewählter Standarabweichungen)
Messreihen instationär: Im Piezometer x wird über ein Jahr jeden Tag gemessen, in Piezometer y hingegen nur 1x pro Monat. Wenn alle Einzelmessungen das gleiche
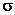 haben (also gleich stark gewichtet werden), wird in der Summe die Grundwassermessstelle x rund 30 Mal stärker gewichtet als die Grundwassermessstelle y (365 Messungen gegenüber 12 Messungen).
haben (also gleich stark gewichtet werden), wird in der Summe die Grundwassermessstelle x rund 30 Mal stärker gewichtet als die Grundwassermessstelle y (365 Messungen gegenüber 12 Messungen).
Abhilfe:  für Piezometer y kleiner wählen.
für Piezometer y kleiner wählen.
Sind die Anteile an der Zielfunktion ausgeglichen, wird die Anzahl der Iterationen auf 10 gesetzt und die inverse Modellierung erneut gestartet.
Ausgabedateien
out.inv
Datei mit Verlauf der Kalibrierung (gemessene/berechnete Werte/Zielfunktion) sowie den K-Werten und Leakagefaktoren im Format der Modelldatei. Die iterierten K-Werte können über Attribute  Modelldaten/Berechnungsergebnisse importieren… direkt auf dem Attribut KWER abgelegt werden.
Modelldaten/Berechnungsergebnisse importieren… direkt auf dem Attribut KWER abgelegt werden.
Invpar.csv:
Datei im Format der Parameterdatei (Achtung: Semikolon als Spaltentrenner, Punkt als Dezimaltrenner). In Spalte D sind die berechneten K-Werte aufgelistet. Diese Datei kann umbenannt werden und dann als Inputdatei für einen nächsten Lauf verwendet werden (z.B. mit teilweise festgehaltenen Werten)
Invpar_xy.csv:
Analog invpar.csv, nur sind Koordinaten vorhanden (nicht für LERA!). Die Datei kann zum Beispiel überlagert werden, um die Ergebnisse zu visualisieren
resan_pote.csv, resan_lkno.csv, resan_knot.csv (nur stationär):
Enthält alle Daten zu den Messwerten
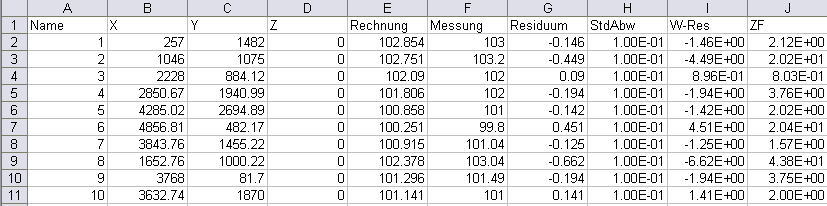
Beispiel der Datei resan_pote.csv
name.csv (nur instationär):
Für jeden Messpunkt (POTE, LKNO, KNOT) wird ein *.csv-Datei mit Gangliniendaten herausgeschrieben. Da hier nicht nach Datenarten unterschieden wird, ist es wichtig, die Namen in der Beobachtungsdatei (*.obs) eindeutig zu definieren (nicht ein Potential namens X1 und eine Mengenmessung namens X1. Besser: h_x1 und q_x1)
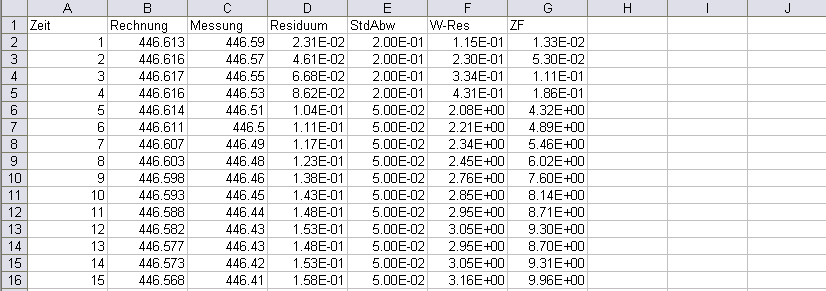
Beispiel einer instationären Ausgabedatei name.csv
